高彩霞研究组在引导编辑研究中取得新进展
发布时间:2021.04.16
新型基因编辑系统引导编辑(prime editing,PE)可以在基因组的靶向位点实现任意类型的碱基替换、小片段的精准插入与删除,这种灵活的、可设计的遗传修饰能力使其在基础研究、人类疾病治疗和农作物育种中潜力巨大。该系统由nCas9(H840A)融合逆转录酶(reverse transcriptase, RT)和pegRNA(prime editing guide RNA)组成,在pegRNA的引导下,nCas9切割非靶标链产生缺刻,释放出可与其PBS序列结合的游离单链,从而起始逆转录酶对RT模板的逆转录过程,然后通过DNA修复将RT模板上对应的碱基改变引入基因组。脱靶效应是基因组编辑技术应用转化时需要解决的一个重要科学问题,然而引导编辑系统在体内是否存在全基因组范围的脱靶尚未知,亟待全面、系统的评估。近日,高彩霞研究组在植物细胞及个体两个水平上对引导编辑系统的脱靶效应进行了深入和系统的评估。
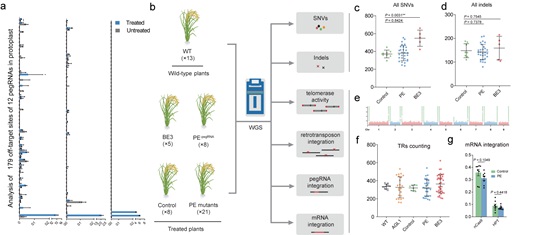
针对引导编辑pegRNA依赖型的脱靶效应,研究人员首先设计了一系列在pegRNA的间隔序列(Spacer)与引物结合位点(PBS)序列不同位置上分别引入数量不同的错配碱基,发现该系统对间隔序列近PAM端或PBS的5’端的错配的容忍程度较低。研究人员进一步对179个潜在的内源脱靶位点的引导编辑情况进行高深度的检测,发现引导编辑系统在水稻内源位点鲜有脱靶编辑。因此,引导编辑系统的pegRNA依赖型的脱靶效应非常低,可通过pegRNA合理设计提升该系统的特异性。
中国科学院遗传与发育生物学研究所高彩霞研究组前期研究表明,胞嘧啶碱基编辑器BE3可能造成不依赖于向导RNA全基因组水平的脱靶效应(Jin et al., Science, 2019)。同时,由于水稻基因组小且再生植株具有相似的遗传背景,可以克服动物全基因组重测序过程中大量的异质细胞序列分析的复杂性难题。因此,研究人员重点评估了引导编辑系统是否造成不依赖pegRNA的全基因组范围的脱靶效应。通过对引导编辑水稻植株的全基因组测序分析,统计了全基因组范围内的碱基替换(single nucleotide variants, SNVs)和小片段插入与删除突变(small insertions/deletions, Indels)数目,发现引导编辑系统的表达不会在基因组内引发额外的SNVs或Indels突变。研究人员还分析了外源过表达含有M-MLV逆转录酶的引导编辑系统是否会干扰细胞内源的逆转录生物学过程。对水稻逆转录转座子OsTos17和端粒区域的拷贝数及保真性分析显示,引导编辑不影响逆转录转座子的转座活性以及端粒酶的活性。此外,对高丰度mRNA及pegRNA序列的逆转录-插入的可能性也进行了分析,同样发现引导编辑系统没有在全基因组范围内产生pegRNA或mRNA序列的随机插入。以上结果表明,引导编辑系统不会造成全基因组范围的不依赖于pegRNA的脱靶效应。
综上所述,引导编辑系统在全基因组范围具有很高的编辑特异性。研究成果于2021年4月15日在线发表在Nature Biotechnology杂志(DOI:10.1038/s41587-021-00891-x)。高彩霞研究组博士生靳帅、林秋鹏、朱子旭以及微生物研究所骆迎峰博士为本文的共同第一作者,高彩霞研究员为本文的通讯作者。该研究得到了国家自然科学基金委、国家重点研发计划、中国科学院战略性先导专项A、中国科学院前沿科学重点研究计划与中国科学院青年创新促进会经费的资助。
 |
|
图:在植物细胞与个体两个水平上对引导编辑系统的特异性进行系统评价。a,水稻原生质体水平分析179个脱靶位点处引导编辑系统的pegRNA依赖型脱靶突变。b,植物个体水平全基因组测序分析引导编辑系统pegRNA不依赖型脱靶突变的实验流程与设计。c,d, control、PE和BE3处理组在基因组内的SNVs(c)与indels(d)数目。e,f,各处理组中含有端粒reads分布及数目比较。g,比较control与PE处理组中Cas9与HPT编码区的reads所占T-DNA区域总reads的比例。 |