曹晓风研究组在植物组蛋白去甲基化酶的招募机制研究中取得新进展
发布时间:2019.05.07
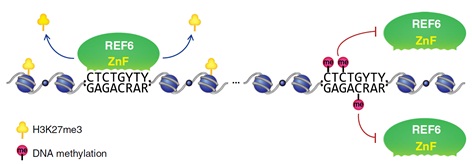
核小体是真核生物染色质的基本单位,由DNA缠绕组蛋白八聚体构成。组蛋白翻译后共价修饰是表观遗传调控的重要方式之一,通过影响染色质的状态而调控基因表达等过程。组蛋白H3第27位赖氨酸的三甲基化修饰(H3K27me3)通过维持基因的沉默状态,在动植物细胞命运决定以及生长发育中发挥重要的调控作用。基因组中特定位点的H3K27me3修饰水平由组蛋白甲基转移酶和去甲基化酶进行动态调控。中国科学院遗传与发育生物学研究所曹晓风研究组前期研究发现,拟南芥组蛋白H3K27me3去甲基化酶REF6/JMJ12能够通过其自身的锌指结构域特异性的识别拟南芥基因组中CTCTGYTY基序从而去除H3K27me3/me2甲基化修饰,调控基因的时空表达水平(Lu, et al. Nature Genetics, 2011; Cui, et al. Nature Genetics, 2016)。进一步研究发现,并非所有的CTCTGYTY基序都能够被REF6识别,REF6更倾向于结合在开放的染色质区域,而不结合异染色质区域,然而这其中的分子机制尚不清楚。DNA甲基化作为一种重要的染色质修饰,广泛分布于异染色质区、常染色质区的转座子区和转录不活跃基因的启动子区,参与异染色质结构维持、转座子沉默和基因转录调控等生物学过程。然而,人们对于植物体内DNA甲基化如何影响组蛋白修饰酶的基因组靶向仍知之甚少。
曹晓风研究组与复旦大学麻锦彪研究组合作,结合染色质组学、结构生物学、生物化学等研究手段,揭示了DNA甲基化抑制REF6对靶位点识别的分子机制。通过生物信息分析REF6的基因组结合位点以及染色质免疫沉淀结合DNA甲基化检测的手段,研究者发现REF6倾向于结合低甲基化水平的CTCTGYTY基序。进一步通过凝胶迁移实验(EMSA)、等温滴定量热法(ITC)以及锌指结构域晶体结构的解析,发现甲基化的DNA基序在体外系统中降低了REF6锌指结构域与DNA结合的亲和力。为了进一步研究植物体内DNA甲基化是否影响REF6与靶基因位点结合,研究者通过染色质免疫沉淀结合高通量测序技术发现,在non-CG甲基化缺失的drm1 drm2 cmt2 cmt3 四突变体中REF6在non-CG甲基化下降的染色质区域会发生异位结合,其大多数位点位于常染色质区中的短TE中间或与之相邻。该研究揭示了DNA甲基化是调控组蛋白去甲基化酶REF6在基因组中靶向的重要因素,为深入开展组蛋白修饰酶类在染色质上的定位和作用机制开拓了思路。
该项研究成果于2019年5月2日在Nature Communications在线发表(DOI:10.1038/s41467-019-10026-1)。曹晓风研究组已毕业博士生邱琦、博士后梅海亮、副研究员邓娴、博士研究生何凯璇和复旦大学麻锦彪组已毕业博士生吴柏星为本文的共同第一作者。中科院遗传发育所曹晓风研究员与复旦大学生命科学学院麻锦彪教授为共同通讯作者。该研究工作由国家自然科学基金委、中国科学院、科技部、中科院青年创新促进会、植物基因组学国家重点实验室等提供经费支持。
 |
|
DNA甲基化抑制REF6结合CTCTGYTY基序模式图 |