梁承志研究组开发高质量基因组组装软件
2019-11-26
2019年11月25日,中国科学院遗传与发育生物学研究所梁承志研究组开发的高质量基因组组装软件HERA在Nature Communications在线发表(DOI:10.1038/s41467-019-13355-3)。论文题目为“Assembly of chromosome-scale contigs by efficiently resolving repetitive sequences with long reads”。
高质量基因组序列对于研究一个物种基因组的结构、功能、进化、基因定位和克隆等都至关重要。目前单分子测序技术的发展,已使得构建高质量基因组草图越来越容易。然而,这些草图序列仍然存在着由于组装序列碎片化而导致的多种错误,比如不完整的基因序列、排列到染色体上之后的片段遗漏、排列顺序错误和方向错误等。这些错误对于利用这些基因组所做的很多研究会造成不便或误导。
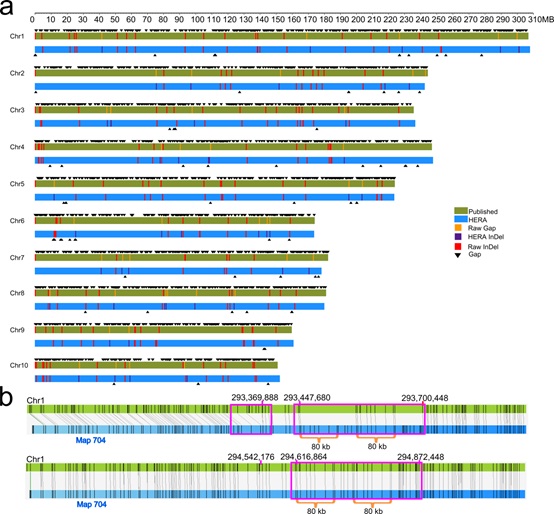
梁承志组多年来通过结合单分子测序和光学图谱及HiC等技术构建高质量基因组,已完成多个植物基因组的组装。最近在前期工作的基础上开发了一个利用单分子测序长片段进行基因组复杂区域组装的新方法HERA。在现有软件组装的基础上,HERA能够大大改进基因组序列的连续性并减少了组装错误。通过对水稻基因组进行测试发现,HERA将水稻中的绝大部分重复序列包括复杂的长串联重复序列都正确地组装了出来。在玉米、苦荞和人基因组中与已发表版本进行对比,玉米的Contig N50从1.3 Mb提升至61.2Mb,人的Contig N50从8.3 MB提升至54.4 MB,苦荞基因组Contig N50达到了27.85 Mb。在玉米B73参考基因组中填补了大量以前没有组装出的序列,校正了多处染色体上序列位置或方向错误,并增加了一些以前丢失的多个重要基因。苦荞中全基因组8条染色体共只由20个Contig组成,其中一条染色体是一个Contig,展示了利用现有常规技术条件构建几乎完整的基因组的潜力。HERA跟已有基因组组装软件CANU等非常互补,预期二者的整合将会产生新的软件,大大提高基因组组装的效率。目前,由于单分子测序价格的下降,组装一个与日本晴质量相当或更好的水稻参考基因组的成本已降到了3万元以下。结合单分子测序、BioNano和Hi-C数据,目前可以很低的成本得到绝大多数物种的高质量参考基因组。对于功能基因组研究来说,高质量基因组序列的获取已不再是一个瓶颈,这预示着后基因组时代在多数物种中的全面到来。
论文第一作者为梁承志研究组博士生杜会龙,通讯作者为梁承志研究员。软件开发得到了基因组分析平台的大力支持和帮助。该研究得到了中国科学院战略性先导科技专项(A)“分子模块设计育种创新体系”等项目的资助。
|
|
|
图a: HERA组装基因组跟玉米参考基因组B73 RefGen_v4的比较。全基因组中序列缺口由2523个减少到了76个。 |
|
图b: 玉米参考基因组中缺失或多余的序列(上图)经HERA改进后(下图)被正确地填补或移除。 |
|