
| 姓 名: | 屠强 |
|---|---|
| 职 称: | 研究员 |
| 职 务: | |
| 电话/传真: | |
| 电子邮件: | qtuATgenetics.ac.cn |
| 实验室主页: | http://tulab.genetics.ac.cn |
| 研究方向: | 系统生物学;发育生物学;基因网络;青鳉鱼;斑马鱼。 |
简历介绍:
工作经历
2014 - 至今 中科院遗传发育所,研究员
2013 - 2014 美国加州理工学院,Member of the Professional Staff (校级学术委员会批准)
2010 - 2013 美国加州理工学院,Senior Research Fellow (Voting Faculty) (校级学术委员会批准)
2004 - 2010 美国加州理工学院,博士后
2003 - 2004 中科院蛋白组分析中心,研究助理
学习经历
2003 博士 中科院生化细胞所
2000 硕士 复旦大学
1997 学士 安徽师范大学
研究领域:
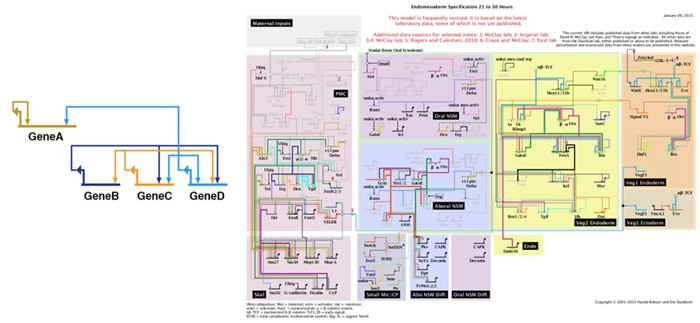

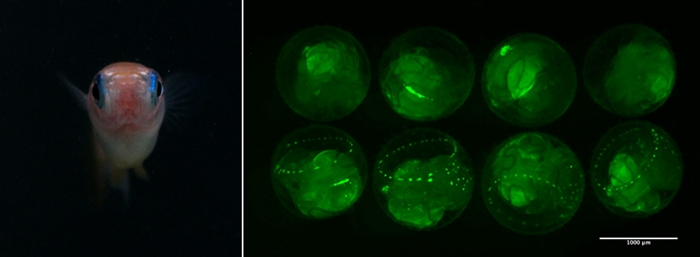
社会任职:
获奖及荣誉:
承担科研项目情况:
代表论著: