田志喜研究组解析大豆三维基因组遗传多样性
发布时间:2023.01.20
染色质的高级结构是基因组中顺式作用元件发挥功能的先决条件,其在基因表达调控中发挥着重要作用。在真核生物中,三维基因组的组织呈现出层次模式,染色质可以在多个层次上划分为不同的结构和功能单位,如染色质疆域(Chromosome Territory, CT)、A/B区室(A/B compartment)、拓扑关联结构域(Topologically Associated Domain, TAD)和染色质环(Chromatin Loop)等。在哺乳动物研究中,胚胎发育中三维基因组的动态变化已有了较多报道。但是,在植物中,尤其在高等植物中,关于三维基因组遗传多样性的深入研究尚未开展。基因组变异和三维基因组变异的关系、三维基因组变异在作物驯化中的效应等诸多科学问题迄今为止仍知之甚少。
中国科学院遗传与发育生物学研究所田志喜研究组根据前期进行基因组重头组装的27份大豆种质材料(Cell, 2020),利用高通量染色质构象捕获(High-through chromosome conformation capture, Hi-C)技术,获得高质量的三维基因组数据。为了调查大豆不同种质中三维基因组的保守性和变异性,研究团队利用泛组学的方法,构建了大豆的泛三维基因组。结果显示,A/B区室在大豆种质间是相对保守的,A/B区室的变异与基因组特征密切相关。与A/B区室不同,TAD边界在27份大豆种质中呈现出更高水平的变异。进一步研究揭示,Non-LTR反转座子(LINE元件和SINE元件),在大豆TAD边界附近呈现富集状态,表明其在植物TAD边界维持中具有重要功能;Gypsy转座元件和卫星重复序列,特异性富集在私有TAD边界附近,表明其在种质特异性TAD边界形成中发挥独特作用。
基因组的结构变异是遗传变异的主要来源。长期以来,由于缺乏高质量的结构变异数据,三维基因组变异和基因组结构变异的关系一直不够明确。研究团队利从基因组组装中获得的高质量结构变异数据,进一步探究了基因组结构变异与三维基因组变异之间的关系。研究显示,在不同类型结构变异中,存在与缺失变异(Presence and Absence Variation, PAV)对三维基因组变异起到了最关键的作用。关联分析表明Gypsy元件和卫星重复序列,在形成私有TAD边界的结构变异中,含量显著升高。这些结果证实了转座元件通过驱动结构变异,从而重塑三维基因组的进化路线。为了探究了三维基因组多样性和基因表达的关系,研究团队从多个水平验证了三维基因组和基因表达的相关性。此外,研究团队还从作物驯化的角度,探究了野生种、地方品种和现代栽培品种中,三维基因组的选择历程。研究表明,三维基因组的选择,主要发生在驯化阶段而非改良阶段,这种选择重塑了基因表达调控的信息,最终导致大豆驯化改良中的基因表达变化。
该研究成果以“Pan-3D genome analysis reveals structural and functional differentiation of soybean genomes”为题,于2023年1月19日在线发表于《Genome Biology》杂志(DOI:10.1186/s13059-023-02854-8)。中科院遗传发育所在读博士生倪令斌为本论文第一作者,田志喜研究员为论文的通讯作者。此外,中科院遗传发育所副研究员张敏、刘书林、申妍婷,博士后刘羽诚、刘腾飞、马新、梁前进,在读学生杨霄月、王钊,以及北京市农林科学院王正研究员也参与了此项研究。该研究得到了国家重点计划研发项目、中国科学院战略重点研究计划、国家自然科学基金等项目的资助。
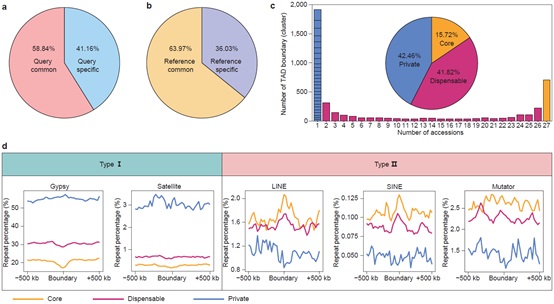
图:大豆种质中TAD边界的泛三维基因组和转座元件分布模式