鲁非研究组完成基于三代测序技术的基因组结构变异检测基准测试
发布时间:2024.09.11
结构变异(Structural Variation)广泛存在于植物基因组中,在基因表达调控、表型建成和适应性进化等方面发挥着关键作用。由于结构变异跨度大、结构复杂等特性,结构变异的精准检测极具挑战性。近些年来,三代测序的发展极大地提升了测序的长度和准确性,这也为结构变异的全基因组精准检测提供了契机。然而,主流的结构变异分析算法和软件多为人类基因组设计和开发,对于复杂植物基因组的适用性尚未评估。因此,开展植物基因组结构变异检测算法的基准测试对揭示结构变异的作用机制具有重要意义。
2024年9月6日,中国科学院遗传与发育生物学研究所鲁非研究组在The Plant Journal在线发表题为“Structural variation discovery in wheat using PacBio high-fidelity sequencing”的研究论文。该论文选取异源六倍体面包小麦及其祖先供体为研究对象,利用PacBio高保真(HiFi)测序数据对三代测序比对算法和结构变异检测算法开展基准测试(图)。结果显示,对于缺失变异(Deletion),结构变异检测软件是检测准确性(F-score)的主要影响因素,可解释准确性总方差的87.73%;而对于插入变异(Insertion),三代测序数据比对软件和结构变异检测软件对检测准确性都有较大贡献,总方差占比分别为38.25%和49.32%。在三代数据比对软件中,Winnowmap2和NGMLR分别适用于检测缺失变异和插入变异,而结构变异检测软件SVIM检测缺失变异和插入变异表现最佳。上述检测软件和比对软件的组合是目前小麦结构变异检测的最佳方法。此外,本研究证实了低覆盖度PacBio HiFi(0.3X)三代测序数据同样能够精准检测基因组结构变异。
该研究提供了目前小麦基因组检测结构变异的最优分析流程,并证明了低覆盖度PacBio HiFi三代测序检测结构变异的能力,为大规模群体的结构变异研究提供了理论与技术支持。鲁非研究组博士生张治梁和张吉瑾为论文共同第一作者,鲁非研究员、尹长斌助理研究员为共同通信作者。该研究得到了国家重点研发计划、国家自然科学基金、中国科学院战略性先导科技专项、海南崖州湾种子实验室“揭榜挂帅”和植物细胞与染色体工程国家重点实验室开放课题等项目的资助。
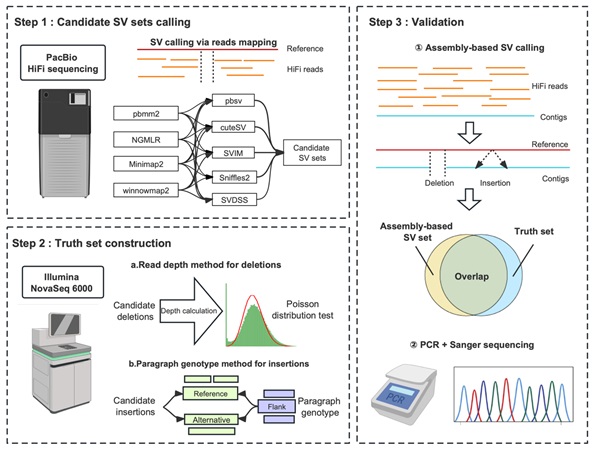
图:基因组结构变异检测算法基准测试概览